Cell:新研究揭示了一种驱动侵袭性骨肉瘤的新机制,并提出一种预测骨肉瘤预后的新型生物标志物
来源:生物谷原创 2025-01-24 11:38
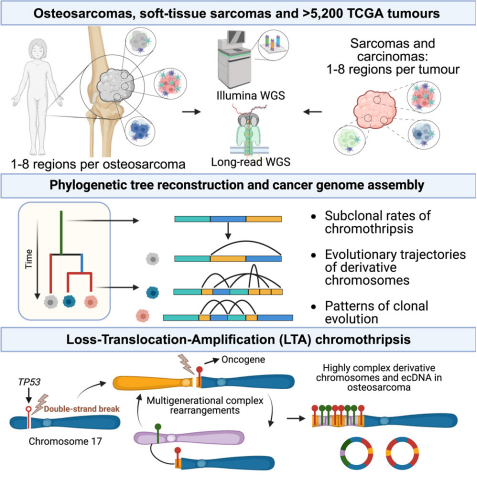
这项新的研究使用长读测序分析了每个骨肉瘤肿瘤的多个区域。这种方法对于确定LTA染色体碎裂机制和发现癌细胞中重排的染色体随着癌症的进展继续获得额外的异常至关重要。
骨肉瘤是一种侵袭性癌症,最常见于10岁至20岁的儿童和年轻人,正值骨骼快速生长时期。虽然罕见,但它对年轻人及其家人产生了重大影响,因为治疗可能需要手术或截肢。
骨肉瘤也有可能扩散到其他器官,最常见的是肺部。由于骨肉瘤在基因组上非常复杂,因此很难确定是什么基因突变导致了这种疾病。因此,在过去的40年里,治疗选择几乎没有进展。

在一项新的研究中,来自EMBL-EBI、伦敦大学学院、皇家国家骨科医院和英格兰基因组学研发实验室的研究人员解决了导致骨肉瘤肿瘤侵袭性发展和进化的基因组重排的驱动因素之谜。通过分析骨肉瘤患者的最大全基因组数据集,他们发现了一种新的称为LTA(loss-translocation-amplification)染色体碎裂的突变机制,这种机制存在于大约50%的高级别骨肉瘤病例中。
相关研究结果于2025年1月14日在线发表在Cell期刊上,论文标题为“Ongoing chromothripsis underpins osteosarcoma genome complexity and clonal evolution”。
这一发现解释了使这种肿瘤类型如此具有侵袭性的独特生物学,以及在骨肉瘤癌细胞中观察到的高水平基因组不稳定性。这项研究还提供了一种预后生物标志物——癌细胞中可以帮助预测患者预后的生物学特征,这可能用于预测这种疾病的可能过程。
论文共同通讯作者、EMBL-EBI组长Isidro Cortes Ciriano说,“多年来,我们已经知道骨肉瘤细胞具有人类癌症中最复杂的基因组,但我们无法解释其背后的机制。通过研究每个肿瘤不同区域的遗传异常,并使用让我们读取长片段DNA的新技术,我们能够了解染色体是如何断裂和重新排列的,以及这如何影响骨肉瘤的疾病进展。”
大规模基因组分析
这项新的研究使用长读测序分析了每个骨肉瘤肿瘤的多个区域。这种方法对于确定LTA染色体碎裂机制和发现癌细胞中重排的染色体随着癌症的进展继续获得额外的异常至关重要。这有助于肿瘤逃避治疗。
这些作者还分析了来自多种癌症类型的5300多份肿瘤的全基因组测序数据。通过这一更广泛的分析,他们发现,各种癌症中出现非常复杂的染色体异常是因为受染色体碎裂影响的染色体高度不稳定。
这一发现对多种癌症类型的治疗具有重要意义,表明骨肉瘤进展中复杂染色体的基因组不稳定性也与其他癌症有关。
论文共同第一作者、EMBL-EBI前博士后研究员Jose Espejo Valle-Inclun说,“我们对不同肿瘤类型的额外分析表明,受复杂基因组重排影响的染色体在其他癌症中也很常见且不稳定。这对我们对癌症发展的总体理解产生了巨大影响,突出了投资于探索这些机制的研究的重要性。”
共同努力
这项研究使用了来自十万基因组计划(100,000 Genomes Project)的数据,其中十万基因组计划是一项由英格兰基因组学研发实验室和英格兰国家医疗服务体系领导的开创性研究,对受罕见疾病或癌症影响的英国国家医疗医疗服务体系患者的全基因组进行了测序。
通过分析大量骨肉瘤患者的基因组数据,这些作者发现约50%的儿童和成人高级别骨肉瘤中存在LTA染色体碎裂。然而,它很少发生在其他癌症类型中,因此突出了对罕见癌症进行大规模分析以确定支持其进化的独特突变的必要性。
英格兰基因组学研发实验室测序研发总监Greg Elgar说,“这些发现大大有助于我们更好地理解是什么驱动了这种侵袭性骨癌的发展,以及它如何在患者体内发展。随着时间的推移,这些新的见解可能通过更有针对性的护理为患者带来更好的治疗选择和结果。”
预测预后
预测骨肉瘤患者的预后,仍然是一个尚未满足的主要需求。作为这项研究的一部分,这些作者还提出了一种新的骨肉瘤预后生物标志物:杂合性缺失(loss of heterozygosity, LOH)。当一个基因组区域的一个拷贝丢失时,就会发生LOH。在骨肉瘤中,整个基因组存在较高的LOH预示着较低的生存几率。
论文共同通讯作者、伦敦大学学院教授Adrienne Flanagan说,“这种生物标志物可能帮助我们识别那些不太可能从治疗中受益的患者,这些治疗可能会产生非常不愉快的效果,并且患者很难忍受。这对于为患者提供更量身定制的治疗,并帮助避免有毒疗法的不必要影响,具有不可估量的价值。”(生物谷 Bioon.com)
参考资料:
Jose Espejo Valle-Inclan et al. Ongoing chromothripsis underpins osteosarcoma genome complexity and clonal evolution. Cell, 2025, doi:10.1016/j.cell.2024.12.005.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


